DNA去甲基化是植物体内一种重要的去除表观遗传修饰标记5-meC的化学反应过程,在维持基因组DNA甲基化的动态平衡、防止基因沉默过程中发挥着至关重要的作用。该过程主要由去甲基化酶ROS1、DEMETER等介导,但详细的分子调控机制仍然很大程度上未知。近年来,虽然已有一些遗传筛选系统被用于鉴定DNA去甲基化通路的新蛋白,但这些系统存在着特异性不强、表型鉴定较难、图位克隆效率不高、鉴定出的蛋白因子的种类集中在几类蛋白等问题,难以发现更多的调控这一通路的新蛋白组分。
近日,国际知名植物分子生物学期刊“Plant Physiology and Biochemistry” (欧洲植物生物学联合会的会刊之一; 5-Year IF: 6.4) 在线发表了南京农业大学生命科学学院腊红桂教授课题组的新科研成果:“Establishment of a forward genetic screening system to discover new proteins associated with DNA demethylation pathway”。该研究成功地构建了一种基于HTC2-pUbq10-Ω-Kozak::LUC (H2UL) 载体的新型高效正向遗传筛选系统,并利用该系统筛选并成功图位克隆了2个直接参与ROS1介导的DNA去甲基化通路的已知基因DML2和APE1L,和一个全新的染色质调控基因RGS3。
该研究团队构建了一个包含内源异染色质序列 (HTC2)、拟南芥内源强启动子 (pUbq10)、Ω-Kozak增强子串联序列和荧光素酶 (LUC) 报告基因的载体,将其转入拟南芥后,获得了发光强烈且稳定的H2UL-1转基因株系。当将ros1-4突变引入该株系后,LUC基因的表达发生显著沉默。全基因组DNA甲基化测序分析发现,LUC基因沉默并非源自于启动子区域的高度甲基化,而是源自于LUC基因下游的终止子 (TerLUC) 上发生的高度甲基化,这一发现增强了对基因沉默机制的新认识。
研究表明,H2UL-1/ros1-4植株中TerLUC的甲基化依赖于RNA-directed DNA Methylation (RdDM) 途径。利用H2UL-1株系进行EMS诱变和筛选,研究人员成功分离出LUC荧光减弱型突变体,并通过图位克隆技术鉴定出3个突变基因:dml2、ape1L和rgs3。其中,DML2和APE1L是已知的、直接参与ROS1介导的碱基切除修复 (BER) 途径的关键基因,但从未通过任何遗传筛选体系获得过这2个基因;而RGS3编码一个功能未知的SFMBT-like (SL) 蛋白,推测其作为组蛋白修饰的“阅读器”,通过识别特定组蛋白标记来调控染色质状态,从而影响DNA去甲基化。酵母双杂交实验表明RGS3能与IDM2、IDM3等已知DNA去甲基化通路的蛋白发生相互作用。
该研究成功建立了一个高效、稳定且易于图位克隆的正向遗传筛选系统。使用该系统不仅能够有效地鉴定出已知的参与DNA去甲基化通路的核心组分,更能够发现全新的调控因子/组分。该筛选系统将为深入揭示ROS1介导的DNA去甲基化途径的分子机制奠定坚实的基础。
南京农业大学生命科学学院博士鲁冲为该论文的第一作者,腊红桂教授为通讯作者。该研究得到了国家自然科学基金等项目的资助。
全文链接:[https://doi.org/10.1016/j.plaphy.2025.110527]
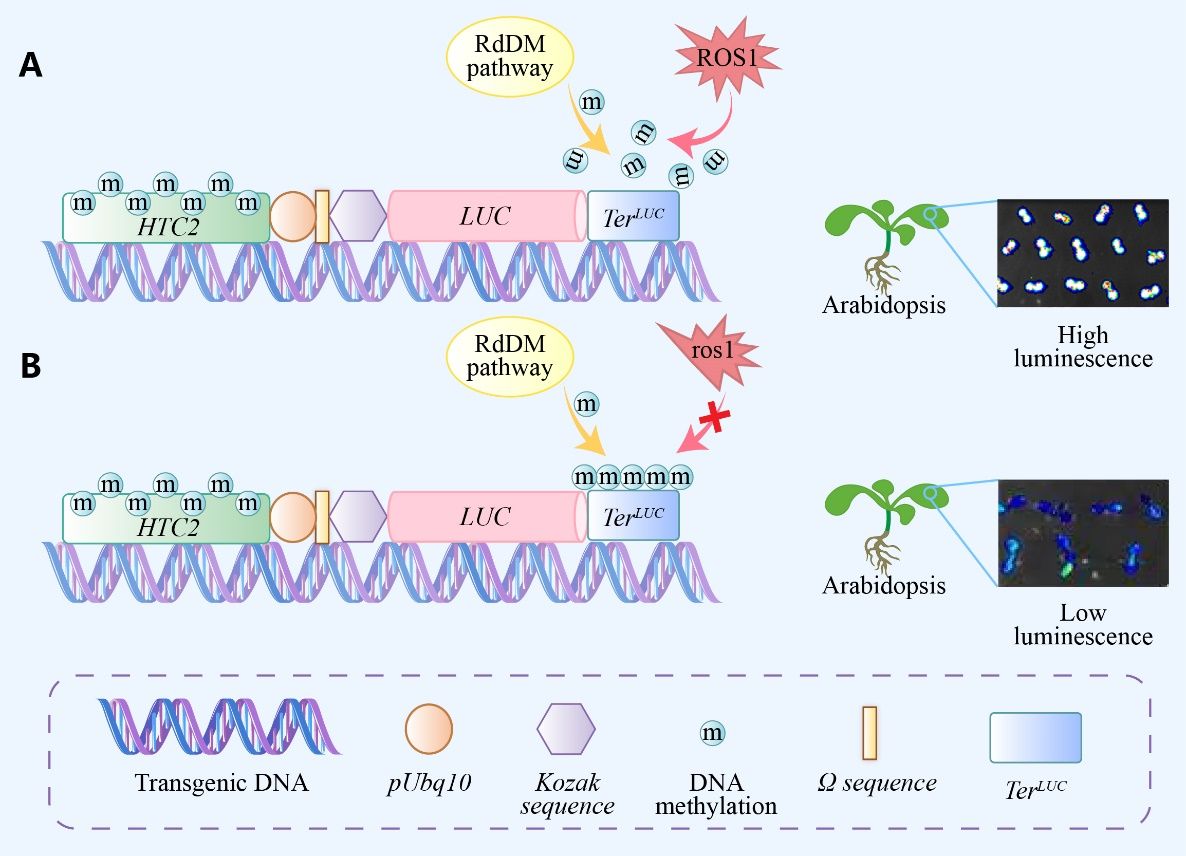
遗传筛选系统的工作原理
