微生物菌群广泛存在于自然界中,在地球上几乎所有的生物地球化学循环中扮演着重要角色。例如,在自然环境中有机污染物的降解往往不是由单一菌株独立完成的,而是需要微生物菌群通过代谢互作来实现。因此,基于微生物菌群内相互作用关系的合成微生物群落,相比单一菌株,能够以更高的效率执行更复杂的任务,在工业、健康和环境领域具有广阔应用前景。然而这些菌群之间复杂的相互作用使得其内在互作机制难以解析,且菌群代谢互作分析和检测工具的不足也使得微生物菌群的实际应用困难重重。
近日,南京农业大学在Nature子刊《Nature Communications》在线发表了题为“Engineering natural microbiomes toward enhanced bioremediation by microbiome modeling”的研究论文。该研究结果为理解和工程化操纵自然微生物菌群以增强其功能开辟了新途径。

重新组装是微生物菌群适应环境变化和干扰的有效策略。因此,通过施加特定的外部选择压力对初始微生物菌群进行驯化,从而促进菌群向特定功能方向重组组装,是一种有效的“自上而下”策略,可以获得具有特定功能的微生物菌群,进而从天然微生物菌群中识别关键微生物(keystone)。在此基础上,采用“自下而上”策略,通过菌群代谢模型技术模拟不同keystone组合的代谢活性,预测最佳菌株组合并进行实验进行验证,最终获得高效合成微生物菌群。据此,研究提出了一种“自上而下”和“自下而上”相结合的方法,可以有效改造自然微生物菌群,设计构建功能增强的合成微生物菌群(图1)。
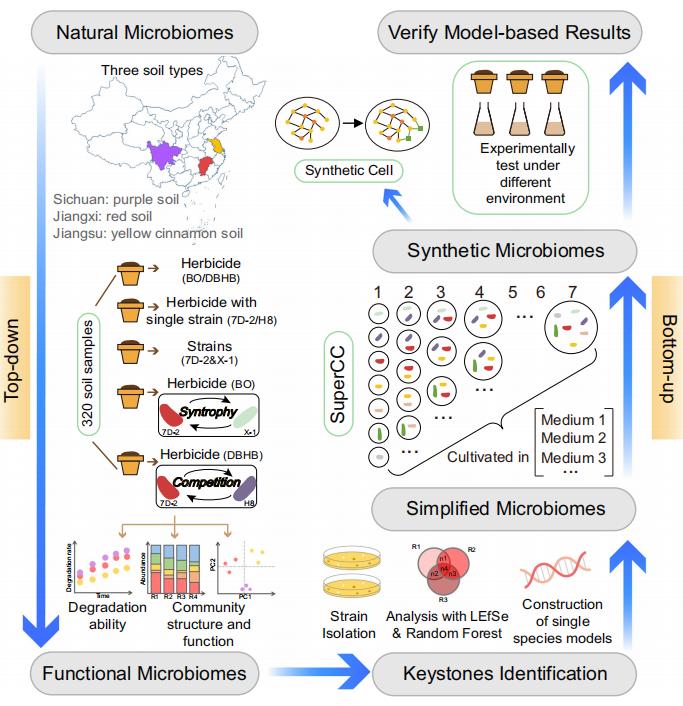
图1 “自上而下”和“自下而上”相结合的合成菌群设计构建方案
为了有效模拟不同菌株组合的代谢活性,研究开发了一个基于微生物组代谢模型技术(microbiome modeling)的最佳功能菌株组合设计工具SuperCC。该工具能够模拟不同营养环境下不同菌群的代谢活性,解析不同菌株间的代谢相互作用。利用SuperCC,研究者基于天然微生物菌群中识别出的18个keystone,构建了用于生物修复的污染物代谢效率增强的合成菌群。本研究的结果强调了代谢相互作用在塑造微生物组功能中的关键作用,并为天然微生物组的工程设计提供了实用的指导。该工具对目标菌株的数量没有限制,因此同时适用于简单和复杂菌群的代谢模型分析。值得注意的时,SuperCC同时提供了一种新的合成细胞设计策略,即鉴定微生物菌群代谢网络中的关键反应,设计优化合成细胞代谢通路,使合成细胞拥有合成菌群的代谢能力(图2)。SuperCC相关原代码已上传至GitHub在线软件源代码托管服务平台,具体内容参见以下网址https://github.com/ruanzhepu/superCC.git。此次在Nature Communications上发表的研究成果是合成菌群研究领域的又一重要研究进展。
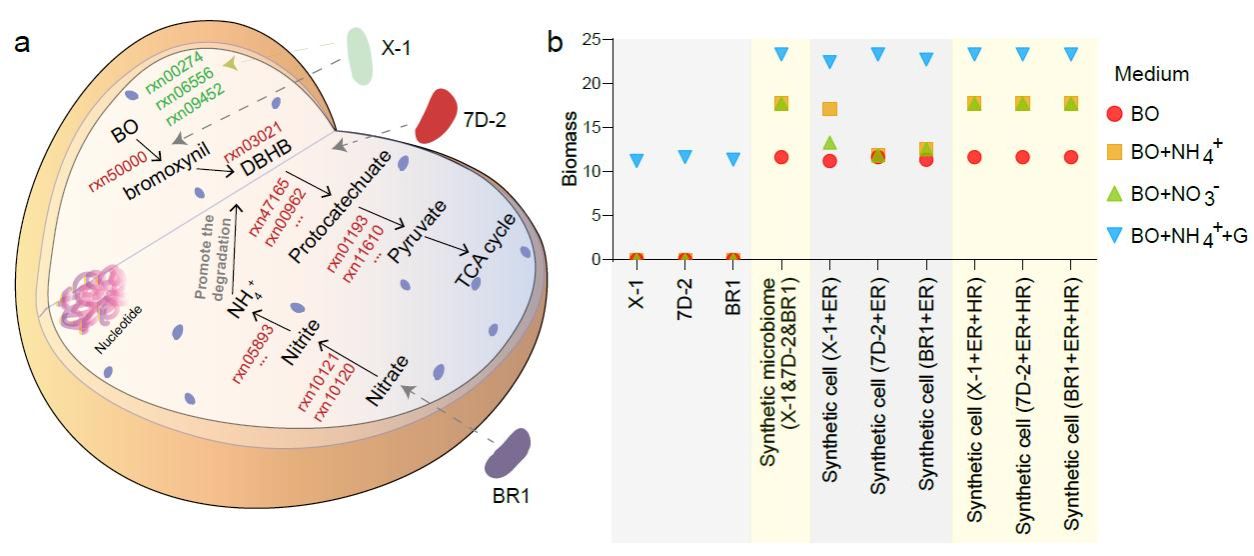
图2 利用SuperCC解析合成菌群关键代谢反应,设计合成细胞代谢通路
已毕业博士阮哲璞(目前为华南农大博士后)和陈凯副教授为该论文共同第一作者,徐希辉教授、蒋建东教授和高彦征教授为论文共同通讯作者,南京农业大学为第一署名单位。研究工作得到了国家重点研发计划、国家自然科学基金等项目的资助。原文链接:https://doi.org/10.1038/s41467-024-49098-z
